腫瘍ゲノム研究部 Department of Cancer Genome Research
メンバー
| 部長(PI) | |||
| 研究員 | Rose Maria Mathew | Rose Maria Mathew | |
| 客員研究員 | 中岡 博史 | Hirofumi Nakaoka | 鹿児島大学医歯学総合研究科データサイエンス分野 Researchmap |
※メールアドレスには@po.kyoundo.jpをつけて下さい
研究内容
卵巣子宮内膜症と正常子宮内膜における遺伝子変異解明
子宮内膜症は子宮内膜に似た組織が子宮以外の組織に生着・増殖し、月経周期に応じて出血や疼痛をきたす疾患で、生殖年齢にある女性の約10%が罹患すると言われています。月経困難症、骨盤痛や不妊症との関連も指摘されており、少子化や女性の社会進出の障害として、社会的損失が大きい疾患です。子宮内膜症が最も多く発症する部位は骨盤腹膜や卵巣です。また、疫学研究により子宮内膜症が特定の組織型の卵巣がん(明細胞腺がん、類内膜がん)の発症リスクになることが報告されています。しかし、卵巣がんの発症母地(前がん病変)とされる卵巣子宮内膜症におけるゲノム変異については明らかにされていませんでした。 そこで、私たちは卵巣子宮内膜症および正常子宮内膜におけるゲノム解析を行い、良性腫瘍である子宮内膜症のみならず正常子宮内膜においても、多数のがん関連遺伝子に変異が存在することを明らかにしました。特に子宮内膜症と関連する卵巣がんで高頻度に認められるKRAS、PIK3CA、FBXW7、PPP2R1A、PIK3R1、ARID1Aの変異が多く認められました。発がんドライバーであるがん関連遺伝子変異が認められたにもかかわらず、これら上皮細胞において病理学的な形態異常は認められませんでした。また、子宮内膜症上皮において、がん関連遺伝子変異、特に卵巣がんで変異頻度の高いKRASおよびPIK3CAに変異を保有する上皮細胞がクローン性に増殖していることが分かりました。 さらに、正常子宮内膜上皮細胞が管状構造(図1)を呈して発達していることに着目し、単一腺管レベルという最小機能単位で体細胞変異プロファイリングを行いました。その結果、正常な子宮内膜から採取した腺管において、KRAS やPIK3CAを含むがん関連遺伝子に体細胞変異が多数検出されました。興味深いことに、各腺管で保有する変異はクローナルな状態に達していましたが、腺管ごとに異なる体細胞変異を保有していました。つまり、子宮内膜組織は、個々に異なる体細胞変異を有する腺管が集合して構成される組織であるため、ゲノムがモザイク状態を呈することが分かりました。これらの結果は、モザイク状ゲノムを呈する子宮内膜が月経血逆流を介して卵巣に生着・増殖する過程で、KRASなどのがん関連遺伝子変異を有する上皮細胞が生存に有利となり、クローナルに増殖することで、子宮内膜症発症に至ることを示唆しています。我々の解析結果は卵巣子宮内膜症発症における月経逆流説をゲノムレベルで支持するものとなりました(図1)。 ヒト正常子宮内膜に存在するがん関連遺伝子変異の機能的影響は未解明です。私たちの研究によって、がん関連遺伝子に体細胞変異を有する細胞クローンが子宮内膜組織において正の選択を受けていることが分かりました。しかし、そのような細胞クローンが存在していても、ほとんどの女性が婦人科がんを発症するには至りません。がん関連遺伝子変異を有する細胞クローンはがん化の危険性という負の側面だけではなく、子宮内膜のように月経周期に応じて再生を繰り返す組織において、増殖能の高い腺管が組織維持に役立っている可能性も考えられます。がん関連遺伝子変異を保有する正常子宮内膜の詳細なゲノム解析を通じて、がん化を抑え、生体を維持する分子機構を解明するための研究に取り組んでいます。 また、子宮内膜だけではなく、肺、胃、食道、皮膚といった他の組織においても、正常細胞における体細胞変異とがん化に関する研究を進めています。様々な正常組織におけるがん関連遺伝子変異の探索・機能的特性を評価することで、組織を越えて共通する分子機構、さらには、組織特有の分子機構を見出すことを目的としています。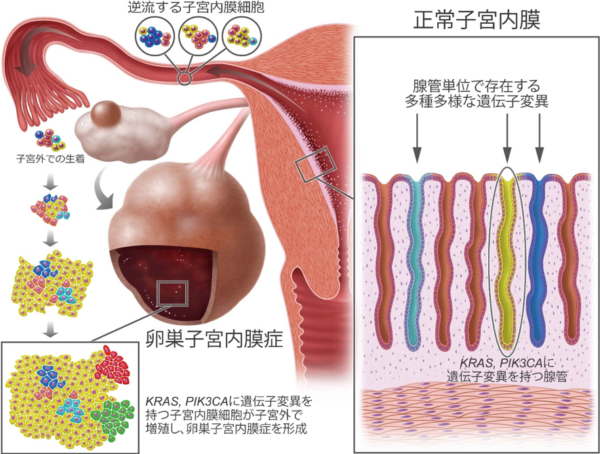
研究業績(過去5年間)
過去5年間の発表論文
- Takahashi K, Yachida N, Tamura R, Adachi S, Kondo S, Abé T, Umezu H, Nyuzuki H, Okuda S, Nakaoka H, Yoshihara K: Clonal origin and genomic diversity in Lynch syndrome-associated endometrial cancer with multiple synchronous tumors: Identification of the pathogenicity of MLH1 p.L582H. Genes Chromosomes Cancer 63: e23231, 2024.
- Nishimura L, Tanino A, Ajimoto M, Katsumura T, Ogawa M, Koganebuchi K, Waku D, Kumagai M, Sugimoto R, Nakaoka H, Oota H, Inoue I: Metagenomic analyses of 7000 to 5500 years old coprolites excavated from the Torihama shell-mound site in the Japanese archipelago. PLoS One 19: e0295924, 2024.
- Akashi H, Yachida N, Ueda H, Yamaguchi M, Yamawaki K, Tamura R, Suda K, Ishiguro T, Adachi S, Nagase Y, Ueda Y, Ueda M, Abiko K, Kagabu M, Baba T, Nakaoka H, Enomoto T, Murai J, Yoshihara K: SLFN11 is a BRCA Independent Biomarker for the Response to Platinum-Based Chemotherapy in High-Grade Serous Ovarian Cancer and Clear Cell Ovarian Carcinoma. Mol Cancer Ther 23: 106-116, 2024.
- Mitsunaga S, Fujito N, Nakaoka H, Imazeki R, Nagata E, Inoue I: Detection of APP gene recombinant in human blood plasma. Sci Rep 13: 21703, 2023.
- Tamura R, Nakaoka H, Yachida N, Ueda H, Ishiguro T, Motoyama T, Inoue I, Enomoto T, Yoshihara K: Spatial genomic diversity associated with APOBEC mutagenesis in squamous cell carcinoma arising from ovarian teratoma. Cancer Science 114: 2145-2157, 2023.
- Toyoda Y, Nakatochi M, Nakayama A, Kawamura Y, Nakaoka H, Wakai K, Matsuo K, Matsuo H; Japan Gout Genomics Consortium: SNP-based heritability estimates of gout and its subtypes determined by genome-wide association studies of clinically defined gout. Rheumatology (Oxford) 62: e144-e146, 2023.
- Banerjee P, Kubo F, Nakaoka H, Ajima R, Sato T, Hirata T, Iwasato T: Spontaneous activity in whisker-innervating region of neonatal mouse trigeminal ganglion. Sci. Rep. 12: 16311, 2022.
- Yoshioka A, Nakaoka H, Fukumoto T, Inoue I, Nishigori C, Kunisada M. The landscape of genetic alterations of UVB-induced skin tumors in DNA repair-deficient mice. Exp. Dermatol. 31: 1607-1617, 2022.
- Rose MM, Dhamodharan S, Revathidevi S, Chakkarappan SR, Mani JG, Shanmugam S, Nakaoka H, Inoue I, Murugan AK, Munirajan AK: High incidence of PI3K pathway gene mutations in South Indian cervical cancers. Cancer Genet. 264-265: 100-108, 2022.
- Iwaya K, Arai H, Takatou N, Morita Y, Ozeki R, Nakaoka H, Sakamoto M, Kouno T, Soma M: A sheet pocket to prevent cross-contamination of formalin-fixed paraffin-embedded block for application in next generation sequencing. PLoS One 17(5):e0266947, 2022.
- Sakaue S, Hosomichi K, Hirata J, Nakaoka H, Yamazaki K, Yawata M, Yawata N, Naito T, Umeno J, Kawaguchi T, Matsui T, Motoya S, Suzuki Y, Inoko H, Tajima A, Morisaki T, Matsuda K, Kamatani Y, Yamamoto K, Inoue I, Okada Y: Decoding the diversity of killer immunoglobulin-like receptors by deep sequencing and a high-resolution imputation method. Cell Genomics 2(3): 100101, 2022.
- Toyoda Y, Nakayama A, Nakatochi M, Kawamura Y, Nakaoka H, Yamamoto K, Shimizu S, Ooyama H, Ooyama K, Shimizu T, Nagase M, Hidaka Y, Ichida K, Inoue I, Shinomiya N, Matsuo H; Japan Gout Genomics Consortium (Japan Gout): Genome-wide meta-analysis between renal overload type and renal underexcretion type of clinically defined gout in Japanese populations. Mol. Genet. Metab. (published online) 2022. doi: 10.1016/j.ymgme.2022.01.100.
- Takahashi M, Hosomichi K, Nakaoka H, Sakata H, Uesato N, Murakami K, Kano M, Toyozumi T, Matsumoto Y, Isozaki T, Sekino N, Otsuka R, Inoue I, Matsubara H: Biased expression of mutant alleles in cancer-related genes in esophageal squamous cell carcinoma. Esophagus 19(2): 294-302, 2022.
- Revathidevi S, Nakaoka H†, Suda K, Fujito N, Munirajan AK, Yoshihara K, Enomoto T, Inoue I†: APOBEC mediated mutagenesis drives genomic heterogeneity in endometriosis. J. Hum. Genet. (6): 323-329, 2022. †Corresponding author.
- Yamaguchi M*, Nakaoka H*, †, Suda K*, Yoshihara K*, †, Ishiguro T, Yachida N, Saito K, Ueda H, Sugino K, Mori Y, Yamawaki K, Tamura R, Revathidevi S, Motoyama T, Tainaka K, Verhaak RGW, Inoue I†, Enomoto T†: Spatiotemporal dynamics of clonal selection and diversification in normal endometrial epithelium. Nat. Commun. 13(1): 943, 2022. *Co-first author. †Corresponding author.
- Jinam TA, Hosomichi K, Nakaoka H, Phipps ME, Saitou N, Inoue I: Allelic and haplotypic HLA diversity in indigenous Malaysian populations explored using Next Generation Sequencing. Hum. Immunol. 83(1): 17-26, 2022.
- Tamura R, Yoshihara K, Matsuo K, Yachida N, Miyoshi A, Takahashi K, Sugino K, Yamaguchi M, Mori Y, Suda K, Ishiguro T, Okuda S, Motoyama T, Nakaoka H, Kikuchi A, Ueda Y, Inoue I, Enomoto T: Proposing a molecular classification associated with hypercoagulation in ovarian clear cell carcinoma. Gynecol. Oncol. 163(2): 327-333, 2021.
- Toyoda Y, Kawamura Y, Nakayama A, Nakaoka H, Higashino T, Shimizu S, Ooyama H, Morimoto K, Uchida N, Shigesawa R, Takeuchi K, Inoue I, Ichida K, Suzuki H, Shinomiya N, Takada T, Matsuo H: Substantial anti-gout effect conferred by common and rare dysfunctional variants of URAT1/SLC22A12. Rheumatology 60: 5224-5232, 2021.
- Sugimoto R, Nishimura L, Nguyen PT, Ito J, Parrish NF, Mori H, Kurokawa K, Nakaoka H, Inoue I: Comprehensive discovery of CRISPR-targeted terminally redundant sequences in the human gut metagenome: Viruses, plasmids, and more. PLoS Comput. Biol. 17: e1009428, 2021.
- Aamer W, Hassan HY, Nakaoka H, Hosomichi K, Jaeger M, Tahir H, Abdelraheem MH, Netea MG, and Inoue I: Analysis of HLA gene polymorphisms in East Africans reveals evidence of gene flow in two Semitic populations from Sudan. Eur. J. Hum. Genet. 29: 1259-1271, 2021.
- Yachida N, Yoshihara K, Suda K, Nakaoka H, Ueda H, Sugino K, Yamaguchi M, Mori Y, Yamawaki K, Tamura R, Ishiguro T, Kase H, Motoyama T, and Enomoto T: Biological significance of KRAS mutant allele expression in ovarian endometriosis. Cancer Sci. 112(5): 2020-2032, 2021.
- Yamaguchi M, Yoshihara K, Suda K, Nakaoka H, Yachida N, Ueda H, Sugino K, Mori Y, Yamawaki K, Tamura R, Ishiguro T, Motoyama T, Watanabe Y, Okuda S, Tainaka K, and Enomoto T: Three-dimensional understanding of the morphological complexity of the human uterine endometrium. iScience. 24(4): 102258, 2021.
- Matsumoto Y, Nagayama H, Nakaoka H, Toyoda A, Goto T, and Koide T: Combined change of behavioral traits for domestication and gene-networks in mice selectively bred for active tameness. Genes Brain Behav. 20(3): e12721, 2021.
- Nishimura L, Sugimoto R, Inoue J, Nakaoka H, Kanzawa-Kiriyama H, Shinoda KI, and Inoue I: Identification of ancient viruses from metagenomic data of the Jomon people. J. Hum. Genet. 66(3): 287-296, 2021.
- Nakakura S, Hosomichi K, Uchino S, Murakami A, Oka A, Inoue I, and Nakaoka H: HLA-B*39:01:01 is a novel risk factor for antithyroid drug-induced agranulocytosis in Japanese population. Pharmacogenomics J. 21(1): 94-101, 2021.
- Revathidevi S, Murugan AK, Nakaoka H, Inoue I, and Munirajan AK: APOBEC: A molecular driver in cervical cancer pathogenesis. Cancer Lett. 496: 104-116, 2021.
- Yesbolatova A, Saito Y, Kitamoto N, Makino-Itou H, Ajima R, Nakano R, Nakaoka H, Fukui K, Gamo K, Tominari Y, Takeuchi H, Saga Y, Hayashi K, and Kanemaki MT: The auxin-inducible degron 2 technology gives superior degradation control in yeast, mammalian cells, and mice. Nat. Commun. 11(1): 5701, 2020.
- Ito J, Kimura I, Soper A, Coudray A, Koyanagi Y, Nakaoka H, Inoue I, Turelli P, Trono D, and Sato K: Endogenous retroviruses drive KRAB zinc-finger protein family expression for tumor suppression. Sci. Adv. 6(43): eabc3020, 2020.
- Yachida N, Yoshihara K, Suda K, Nakaoka H, Ueda H, Sugino K, Yamaguchi M, Mori Y, Yamawaki K, Tamura R, Ishiguro T, Isobe M, Motoyama T, Inoue I, and Enomoto T: ARID1A protein expression is retained in ovarian endometriosis with ARID1A loss-of-function mutations: Implication for the two-hit hypothesis. Sci. Rep. 10(1): 14260, 2020.
- Kantake M, Ikeda N, Nakaoka H, Ohkawa N, Tanaka T, Miyabayashi K, Shoji H, and Shimizu T: IGF1 gene is epigenetically activated in preterm infants with intrauterine growth restriction. Clin. Epigenetics. 12(1): 108, 2020.
- Suda K, Cruz Diaz LA, Yoshihara K, Nakaoka H, Yachida N, Motoyama T, Inoue I, and Enomoto T: Clonal lineage from normal endometrium to ovarian clear cell carcinoma through ovarian endometriosis. Cancer Sci. 111(8): 3000–3009, 2020.
- Hata C, Nakaoka H, Xiang Y, Wang D, Yang A, Liu D, Liu F, Zou Q, Wei L, Zheng K, Inoue I, and You H: Germline mutations of multiple breast cancer-related genes are differentially associated with triple-negative breast cancers and prognostic factors. J. Hum. Genet. 65(7):577-587, 2020.
- Nakayama A*, Nakatochi M*, Kawamura Y*, Yamamoto K*, Nakaoka H*, Shimizu S, Higashino T, Koyama T, Hishida A, Kuriki K, Watanabe M, Shimizu T, Ooyama K, Ooyama H, Nagase M, Hidaka Y, Matsui D, Tamura T, Nishiyama T, Shimanoe C, Katsuura-Kamano S, Takashima N, Shirai Y, Kawaguchi M, Takao M, Sugiyama R, Takada Y, Nakamura T, Nakashima H, Tsunoda M, Danjoh I, Hozawa A, Hosomichi K, Toyoda Y, Kubota Y, Takada T, Suzuki H, Stiburkova B, Major TJ, Merriman TR, Kuriyama N, Mikami H, Takezaki T, Matsuo K, Suzuki S, Hosoya T, Kamatani Y, Kubo M, Ichida K, Wakai K, Inoue I, Okada Y, Shinomiya N, and Matsuo H on behalf of Japan Gout Genomics Consortium (Japan Gout). Subtype-specific gout susceptibility loci and enrichment of selection pressure on ABCG2 and ALDH2 identified by subtype genome-wide meta-analyses of clinically-defined gout patients. Ann Rheum Dis. 79(5):657-665, 2020. *Co-first author.
- Tamura R, Yoshihara K, Nakaoka H, Yachida N, Yamaguchi M, Suda K, Ishiguro T, Nishino K, Ichikawa H, Honma K, Kikuchi A, Ueda Y, Takei Y, Fujiwara H, Motoyama T, Okuda S, Wakai T, Inoue I, and Enomoto T. XCL1 expression correlates with CD8 positive T cells infiltration and PD-L1 expression in squamous cell carcinoma arising from mature cystic teratoma of the ovary. Oncogene. 39(17):3541-3554, 2020.
- Higashino T*, Morimoto K*, Nakaoka H*, Toyoda Y*, Kawamura Y*, Shimizu S*, Nakamura T, Hosomichi K, Nakayama A, Ooyama K, Ooyama H, Shimizu T, Ueno M, Ito T, Tamura T, Naito M, Nakashima H, Kawaguchi M, Takao M, Kawai Y, Osada N, Ichida K, Yamamoto K, Suzuki H, Shinomiya N, Inoue I, Takada T, and Matsuo H. Dysfunctional missense variant of OAT10/SLC22A13 decreases gout risk and serum uric acid levels. Ann. Rheum. Dis. 79(1):164-166, 2020.*Co-first author.
- Zhang S, Kohira Y, Orita H, Ishimine M, Kobayashi T, Chua SMB, Nakaoka H, Inoue I, Hino O, Yokomizo T, Fukunaga T, and Lee-Okada HC. Sensitization of gastric cancer cells to irinotecan by p53 activation. BPB Rep. 2: 130-133, 2019.
- Sugino K, Tamura R, Nakaoka H, Yachida N, Yamaguchi M, Mori Y, Yamawaki K, Suda K, Ishiguro T, Adachi S, Isobe M, Yamaguchi M, Kashima K, Motoyama T, Inoue I, Yoshihara K, and Enomoto T. Germline and somatic mutations of homologous recombination-associated genes in Japanese ovarian cancer patients. Sci. Rep. 9(1):17808, 2019.
- Suda K, Nakaoka H, Yoshihara K, Ishiguro T, Adachi S, Kase H, Motoyama T, Inoue I, and Enomoto T. Different mutation profiles between epithelium and stroma in endometriosis and normal endometrium. Hum. Reprod. 34(10):1899-1905, 2019.
- Hata C, Nakaoka H, Xiang Y, Wang D, Yang A, Liu D, Liu F, Zou Q, Zheng K, Inoue I, and You H. Exploration of intermediate-sized indels by next-generation multigene panel testing in Han Chinese patients with breast cancer. Hum. Genome Var.. 6:51, 2019.
- Kawamura Y*, Nakaoka H*, Nakayama A*, Okada Y*, Yamamoto K*, Higashino T, Sakiyama M, Shimizu T, Ooyama H, Ooyama K, Nagase M, Hidaka Y, Shirahama Y, Hosomichi K, Nishida Y, Shimoshikiryo I, Hishida A, Katsuura-Kamano S, Shimizu S, Kawaguchi M, Uemura H, Ibusuki R, Hara M, Naito M, Takao M, Nakajima M, Iwasawa S, Nakashima H, Ohnaka K, Nakamura T, Stiburkova B, Merriman TR, Nakatochi M, Ichihara S, Yokota M, Takada T, Saitoh T, Kamatani Y, Takahashi A, Arisawa K, Takezaki T, Tanaka K, Wakai K, Kubo M, Hosoya T, Ichida K, Inoue I, Shinomiya N, and Matsuo H. Genome-wide association study revealed novel loci which aggravate asymptomatic hyperuricaemia into gout. Ann. Rheum. Dis. 78(10):1430-1437, 2019. *Co-first author.
- Suda K, Nakaoka H, Hata C, Yahata N, Isobe M, Kameyama H, Wakai T, Motoyama T, Inoue I, Yoshihara K, and Enomoto T. Concurrent isolated retroperitoneal HGSC and STIC defined by somatic mutation analysis: a case report. Diagn. Pathol. 14(1):17, 2019.
- Hirata J, Hosomichi K, Sakaue S, Kanai M, Nakaoka H, Ishigaki K, Suzuki K, Akiyama M, Kishikawa T, Ogawa K, Masuda T, Yamamoto K, Hirata M, Matsuda K, Momozawa Y, Inoue I, Kubo M, Kamatani Y, and Okada Y. Genetic and phenotypic landscape of the major histocompatibility complex region in the Japanese population. Nat. Genet.51(3):470-480, 2019.
それ以前の代表的な発表論文
- Suda K*, Nakaoka H*, Yoshihara K, Ishiguro T, Tamura R, Mori Y, Yamawaki K, Adachi S, Takahashi T, Kase H, Tanaka K, Yamamoto T, Motoyama T, Inoue I, and Enomoto T. Clonal expansion and diversification of cancer-associated mutations in endometriosis and normal endometrium. Cell Rep. 24(7):1777-1789, 2018. *Co-first author.
- Nakaoka H, Gurumurthy A, Hayano T, Ahmadloo A, Omer WH, Yoshihara K, Yamamoto A, Kurose K, Enomoto T, Akira S, Hosomichi K, and Inoue I. Allelic imbalance in regulation of ANRIL through chromatin interaction at 9p21 endometriosis risk locus. PLOS Genet. 12(4): e1005893, 2016.
- Matsuo H*, Yamamoto K*, Nakaoka H*, Nakayama A*, Sakiyama M*, Chiba T, Takahashi A, Nakamura T, Nakashima H, Takada Y, Danjoh I, Shimizu S, Abe J, Kawamura Y, Terashige S, Ogata H, Tatsukawa S, Guang Y, Okada R, Morita E, Naito M, Tokumasu A, Onoue H, Iwaya K, Ito T, Takada T, Inoue K, Kato Y, Nakamura Y, Sakurai Y, Suzuki H, Kanai Y, Hosoya T, Hamajima N, Inoue I, Kubo M, Ichida K, Ooyama H, Shimizu T, and Shinomiya N. Genome-wide association study of clinically defined gout identifies multiple risk loci and its association with clinical subtypes. Ann. Rheum. Dis. 75(4): 652-659, 2016. *Co-first author.
- Nakaoka H and Inoue I. Distribution of HLA haplotypes across Japanese Archipelago: similarity, difference and admixture. J. Hum. Genet. 60(11): 683-690, 2015.
- Nakaoka H, Tajima A, Yoneyama T, Hosomichi K, Kasuya H, Mizutani T, and Inoue I. Gene expression profiling reveals distinct molecular signatures associated with the rupture of intracranial aneurysm. Stroke. 45(8): 2239-2245, 2014.
- Nakaoka H, Takahashi T, Akiyama K, Cui T, Tajima A, Krischek B, Kasuya H, Hata A, and Inoue I: Differential effects of chromosome 9p21 variation on subphenotypes of intracranial aneurysm: site distribution. Stroke. 41(8): 1593-1598, 2010.
- Nakaoka H and Inoue I: Meta-analysis of genetic association studies: methodologies, between-study heterogeneity and winner’s curse. J. Hum. Genet. 54(11): 615-623, 2009.